Impatto clinico del panorama genico e dei percorsi di leucemogenesi nei pazienti anziani con AML trattati con terapia non intensiva
Articolo originale: Jahn E. et al. Clinical impact of the genomic landscape and leukemogenic trajectories in non-intensively treated elderly acute myeloid leukemia patients, Leukemia 2023; 37: 2187–2196.
La classificazione della European LeukemiaNet (ELN) non considera i pazienti anziani trattati in modo non-intensivo. Gli autori hanno sequenziato il DNA di 263 geni in 604 pazienti arruolati nello studio di fase III ASTRAL-1 (guadecitabina vs decitabina o azacitidina o citarabina a basse dosi).
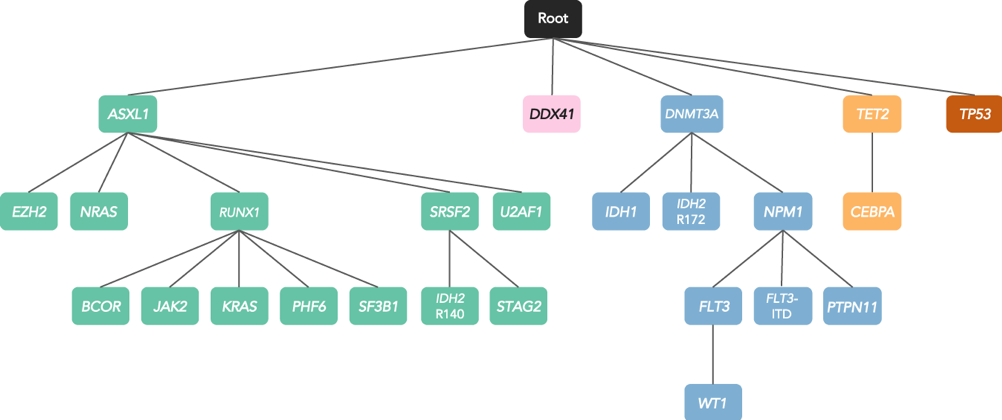
Sono state analizzate 2985 mutazioni con frequenza allelica ≥4% e alterazioni citogenetiche, analizzandone l’impatto sulla sopravvivenza, la mutua esclusività, la co-occorrenza e l’ordine temporale di acquisizione fino a disegnare l’albero oncogenetico includente cinque gruppi con mutazioni ASXL1, DDX41, DNMT3A, TET2 e TP53 identificate come eventi iniziali che predispongono a eventi aggiuntivi con la genesi di cloni distinti (figura 1).
Figura 1. ASXL1 è associato alle mutazioni genetiche correlate a mielodisplasia. DNMT3A spesso acquisisce NPM1 prima delle alterazioni di signaling, ultimi eventi della leucemogenesi (NRAS, KRAS, JAK2, FLT3, PTPN1), e a volte acquisisce IDH1/2. TET2 può acquisire CEBPA. Le mutazioni DDX41 (n=33, 5,5%), spesso germinali (n=20, 3,3%), e quelle di TP53 non risultano associate ad altre mutazioni.
Sono stati individuati 5 gruppi e 3 sottogruppi sulla base delle analisi di clustering/correlazione tra alterazioni molecolari e citogenetiche presenti in >1% delle AML:
- Classe 1 (49%, n= 279) con mutazioni genetiche correlate alla mielodisplasia e alterazioni cromosomiche correlate alla mielodisplasia, associate spesso alle mutazioni NRAS e KRAS.
- Classe 2 (25%, n=142) con cariotipo complesso, (93%, n = 131), −5/del(5q), −7/del(7q), -17/del(17p)/abn(17p) associati alle mutazioni di TP53 (70%, n = 99).
- Classe 3 (17%, n=95) con mutazioni delle AML de novo come NPM1, DNMT3A, FLT3 -ITD, IDH1 e IDH2 R140.
- Classe 4 (n=9) con varie alterazioni cromosomiche strutturali e numeriche.
- Classe 5 (n=11) con IDH2 R172associata a DNMT3A (n=8).
Sottogruppi:
- Alterazioni DDX41 (n=18), associate a un cariotipo normale (16/18).
- Mutazioni CBF (n=6).
- Riarrangiamenti MECOM (EVI1) (n =5).
Il rischio ELN e la classificazione secondo l'International Consensus Classification (ICC) non permettono di identificare nettamente la prognosi. La maggioranza dei pazienti ha rischio ELN 2017/2022 sfavorevole (62%, n=363/73%, n=428), seguiti dal rischio intermedio (21% , n=124/14%, n=85) e dal rischio favorevole (17%, n=101/13%, n=74).
L’analisi multivariata ha identificato età, sesso maschile, punteggio di Eastern Cooperative Oncology Group (ECOG) >2, leucocitosi (log10), mutazioni TP53 (OS mediana 5,1 mesi, HR 1,59), FLT3 -ITD (OS mediana 4,9 mesi, HR 1,7), SRSF2 (OS a 2 anni 8,36%, HR 1,36) come fattori sfavorevoli, mentre DDX41 (OS mediana 27,8 vs 7,1 mesi, HR 0,41; 0,24–0,69) ha prognosi favorevole. NPM1 perde il significato favorevole con HR 0,94 e mediana di OS pari a 6,1 mesi. Quando si valutano le alterazioni citogenetiche il cariotipo complesso è risultato un fattore avverso altamente significativo al posto di TP53.
Diversi modelli sono ugualmente predittivi di sopravvivenza quando confrontati tra loro:
- Variabili cliniche (età, sesso maschile, ECOG>2, leucocitosi, braccio di trattamento) e tutte le mutazioni con VAF>4%.
- ECOG, conta dei globuli bianchi (WBC), mutazioni FLT3-ITD, TP53, e DDX41 (mutualmente esclusive).
- Random survival forest includente variabili cliniche e mutazioni.
- Modello con sole variabili cliniche.
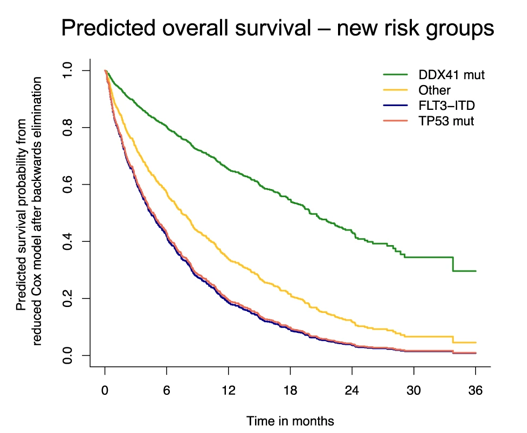
Le mutazioni sono in grado di predire la sopravvivenza, anche dopo aggiustamento per conta leucocitaria mediana (<3,6×109/L) ed ECOG <2, identificando 3 gruppi: favorevole con mutazione DDX41, intermedio senza mutazioni DDX41, TP53 e FLT3-ITD, sfavorevole con mutazioni TP53 o FLT3-ITD (figura 2).
Figura 2. Sopravvivenza e mutazioni aggiustate per conta leucocitaria ed ECOG.
Nelle analisi retrospettive post-hoc dello studio VIALE-A, l'AML sia FLT3 -ITD sia TP53- mutata ha dimostrato di avere prognosi sfavorevole anche se trattata con venetoclax (1 , 2).
La mutazione DDX41 potrebbe essere associata all'efficacia della monoterapia con agenti ipometilanti (HMA) (3) o con HMA e venetoclax. La frequente origine germinale di DDX41, che dà inizio alla leucemia, e la scarsità di lesioni molecolari e citogenetiche concomitanti, suggeriscono che l'AML con DDX41 mutato possa essere un’entità clinico-patologica distinta.
BIBLIOGRAFIA
-
Pollyea DA, Pratz KW, Wei AH, et al. Outcomes in patients with poor-risk cytogenetics with or without TP53 mutations treated with venetoclax and azacitidine. Clin Cancer Res 2022; 28: 5272–5279.
-
Konopleva M, Thirman MJ, Pratz KW, et al. Impact of FLT3 mutation on outcomes after venetoclax and azacitidine for patients with treatment-naïve acute myeloid leukemia. Clin Cancer Res 2022; 28: 2744–2752.
-
Makishima H, Saiki R, Nannya Y, et al. Germ line DDX41 mutations define a unique subtype of myeloid neoplasms. Blood 2023; 141: 534–549.